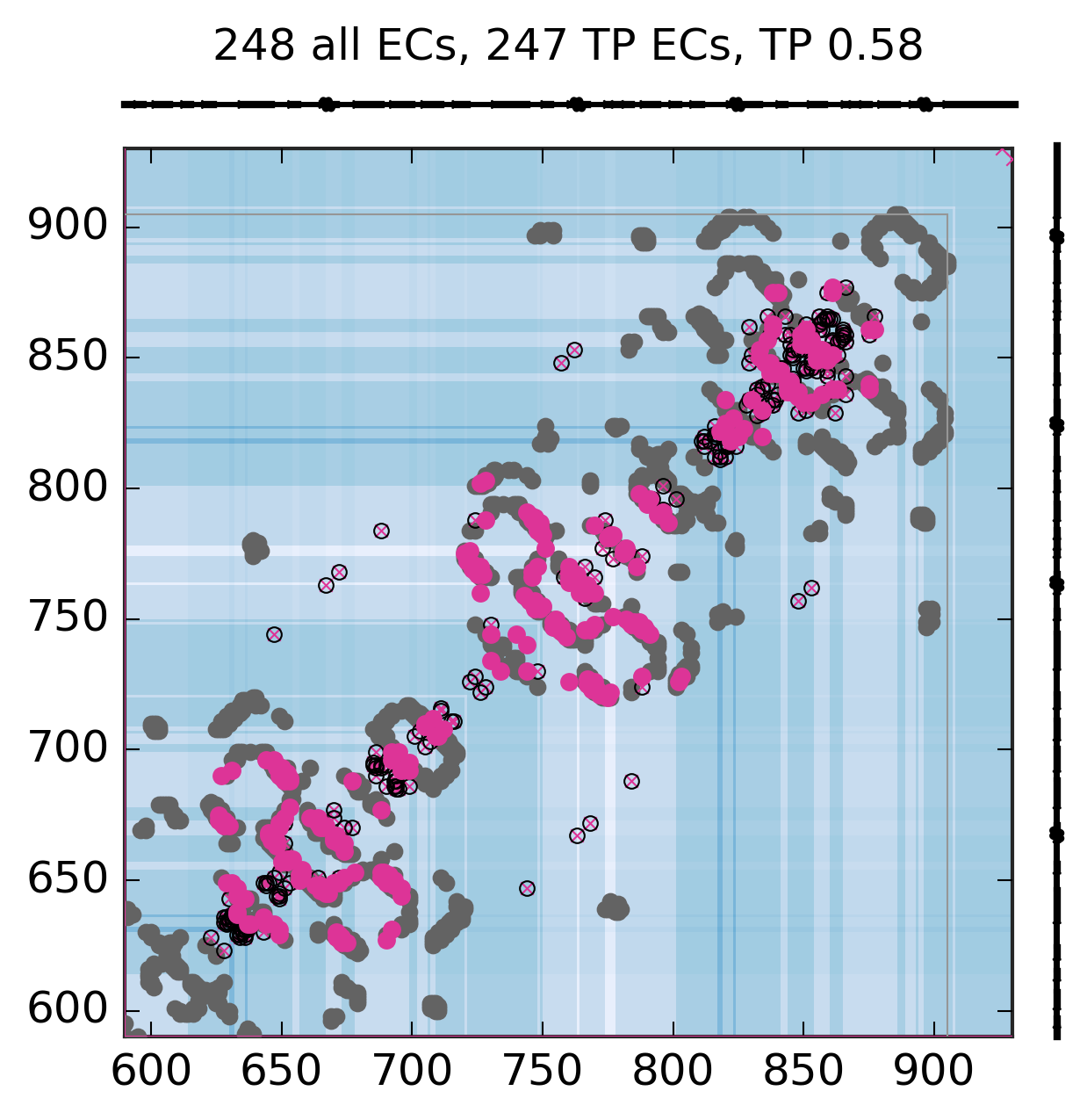
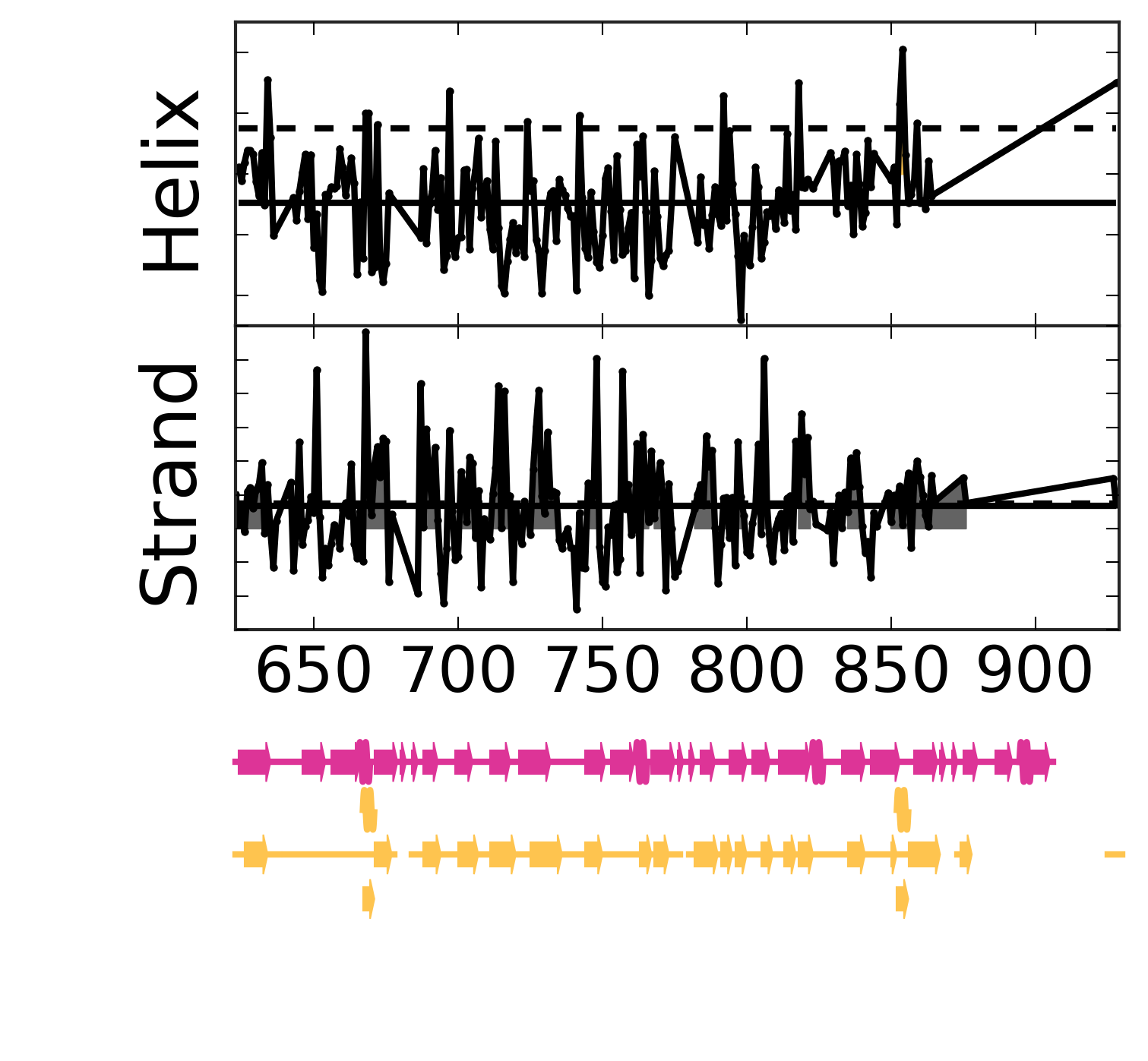
Evolutionary Coupling Analysis
Predicted and experimental contacts
Secondary structure from ECs
EC score distribution and threshold
Top ECs
| Rank |
Residue 1 |
Amino acid 1 |
Residue 2 |
Amino acid 2 |
EC score |
| 1 |
747 |
N |
789 |
D |
0.93 |
| 2 |
749 |
L |
787 |
T |
0.79 |
| 3 |
747 |
N |
754 |
K |
0.77 |
| 4 |
726 |
G |
768 |
Y |
0.71 |
| 5 |
652 |
Y |
689 |
T |
0.71 |
| 6 |
650 |
E |
693 |
L |
0.71 |
| 7 |
748 |
L |
770 |
F |
0.68 |
| 8 |
728 |
K |
788 |
E |
0.68 |
| 9 |
667 |
S |
671 |
T |
0.67 |
| 10 |
648 |
K |
694 |
T |
0.65 |
| 11 |
749 |
L |
754 |
K |
0.65 |
| 12 |
650 |
E |
691 |
E |
0.64 |
| 13 |
666 |
E |
670 |
W |
0.61 |
| 14 |
664 |
L |
672 |
H |
0.61 |
| 15 |
628 |
T |
671 |
T |
0.61 |
| 16 |
695 |
K |
699 |
Y |
0.59 |
| 17 |
816 |
V |
820 |
W |
0.57 |
| 18 |
652 |
Y |
691 |
E |
0.57 |
| 19 |
852 |
N |
856 |
N |
0.56 |
| 20 |
665 |
N |
670 |
W |
0.56 |
| 21 |
647 |
I |
696 |
V |
0.55 |
| 22 |
645 |
T |
668 |
N |
0.55 |
| 23 |
750 |
A |
782 |
I |
0.54 |
| 24 |
648 |
K |
663 |
I |
0.54 |
| 25 |
747 |
N |
787 |
T |
0.53 |
| 26 |
631 |
K |
692 |
E |
0.52 |
| 27 |
647 |
I |
695 |
K |
0.50 |
| 28 |
711 |
L |
715 |
N |
0.49 |
| 29 |
629 |
V |
672 |
H |
0.48 |
| 30 |
837 |
E |
845 |
T |
0.48 |
| 31 |
664 |
L |
674 |
W |
0.47 |
| 32 |
651 |
L |
674 |
W |
0.47 |
| 33 |
849 |
A |
859 |
H |
0.46 |
| 34 |
670 |
W |
674 |
W |
0.46 |
| 35 |
632 |
N |
636 |
N |
0.46 |
| 36 |
857 |
W |
864 |
L |
0.45 |
| 37 |
926 |
K |
930 |
K |
0.45 |
| 38 |
762 |
S |
766 |
W |
0.44 |
| 39 |
725 |
S |
767 |
K |
0.44 |
| 40 |
830 |
R |
851 |
L |
0.44 |
| 41 |
628 |
T |
673 |
T |
0.44 |
| 42 |
649 |
V |
692 |
E |
0.44 |
| 43 |
790 |
H |
794 |
Y |
0.43 |
| 44 |
650 |
E |
657 |
A |
0.42 |
| 45 |
851 |
L |
855 |
N |
0.42 |
| 46 |
751 |
D |
777 |
D |
0.42 |
| 47 |
775 |
K |
781 |
K |
0.42 |
| 48 |
746 |
V |
788 |
E |
0.41 |
| 49 |
705 |
N |
710 |
N |
0.41 |
| 50 |
727 |
E |
767 |
K |
0.40 |
| 51 |
837 |
E |
844 |
A |
0.40 |
| 52 |
725 |
S |
769 |
E |
0.40 |
| 53 |
857 |
W |
861 |
W |
0.40 |
| 54 |
686 |
V |
690 |
V |
0.40 |
| 55 |
750 |
A |
755 |
V |
0.40 |
| 56 |
677 |
L |
688 |
Y |
0.39 |
| 57 |
744 |
V |
791 |
V |
0.39 |
| 58 |
666 |
E |
672 |
H |
0.39 |
| 59 |
760 |
V |
770 |
F |
0.38 |
| 60 |
626 |
S |
673 |
T |
0.38 |
| 61 |
837 |
E |
848 |
T |
0.38 |
| 62 |
812 |
T |
820 |
W |
0.38 |
| 63 |
747 |
N |
757 |
T |
0.38 |
| 64 |
841 |
D |
846 |
G |
0.38 |
| 65 |
853 |
E |
857 |
W |
0.38 |
| 66 |
653 |
Q |
688 |
Y |
0.37 |
| 67 |
688 |
Y |
693 |
L |
0.37 |
| 68 |
653 |
Q |
658 |
T |
0.37 |
| 69 |
760 |
V |
764 |
T |
0.37 |
| 70 |
630 |
T |
634 |
D |
0.37 |
| 71 |
720 |
E |
775 |
K |
0.37 |
| 72 |
835 |
K |
848 |
T |
0.37 |
| 73 |
812 |
T |
816 |
V |
0.37 |
| 74 |
730 |
W |
744 |
V |
0.36 |
| 75 |
630 |
T |
643 |
R |
0.36 |
| 76 |
761 |
T |
766 |
W |
0.36 |
| 77 |
692 |
E |
699 |
Y |
0.36 |
| 78 |
776 |
Y |
781 |
K |
0.36 |
| 79 |
823 |
N |
827 |
D |
0.35 |
| 80 |
633 |
W |
637 |
N |
0.35 |
| 81 |
814 |
A |
818 |
K |
0.35 |
| 82 |
651 |
L |
690 |
V |
0.35 |
| 83 |
627 |
A |
690 |
V |
0.34 |
| 84 |
745 |
S |
790 |
H |
0.34 |
| 85 |
631 |
K |
649 |
V |
0.34 |
| 86 |
861 |
W |
877 |
V |
0.34 |
| 87 |
763 |
E |
767 |
K |
0.33 |
| 88 |
745 |
S |
757 |
T |
0.33 |
| 89 |
845 |
T |
859 |
H |
0.33 |
| 90 |
748 |
L |
784 |
Y |
0.33 |
| 91 |
749 |
L |
785 |
T |
0.33 |
| 92 |
648 |
K |
665 |
N |
0.32 |
| 93 |
820 |
W |
834 |
I |
0.32 |
| 94 |
646 |
E |
665 |
N |
0.32 |
| 95 |
723 |
S |
771 |
K |
0.32 |
| 96 |
836 |
V |
857 |
W |
0.32 |
| 97 |
711 |
L |
716 |
K |
0.32 |
| 98 |
840 |
Q |
844 |
A |
0.32 |
| 99 |
628 |
T |
634 |
D |
0.32 |
| 100 |
832 |
T |
838 |
L |
0.32 |
| 101 |
820 |
W |
825 |
N |
0.32 |
| 102 |
746 |
V |
768 |
Y |
0.32 |
| 103 |
688 |
Y |
784 |
Y |
0.31 |
| 104 |
630 |
T |
671 |
T |
0.31 |
| 105 |
632 |
N |
637 |
N |
0.31 |
| 106 |
749 |
L |
755 |
V |
0.31 |
| 107 |
705 |
N |
709 |
G |
0.31 |
| 108 |
722 |
T |
726 |
G |
0.31 |
| 109 |
726 |
G |
760 |
V |
0.31 |
| 110 |
740 |
R |
744 |
V |
0.31 |
| 111 |
743 |
K |
759 |
D |
0.30 |
| 112 |
686 |
V |
699 |
Y |
0.30 |
| 113 |
686 |
V |
694 |
T |
0.30 |
| 114 |
633 |
W |
638 |
N |
0.30 |
| 115 |
667 |
S |
763 |
E |
0.30 |
| 116 |
839 |
Y |
844 |
A |
0.30 |
| 117 |
760 |
V |
768 |
Y |
0.30 |
| 118 |
672 |
H |
768 |
Y |
0.30 |
| 119 |
777 |
D |
782 |
I |
0.30 |
| 120 |
651 |
L |
688 |
Y |
0.30 |
| 121 |
791 |
V |
796 |
T |
0.30 |
| 122 |
766 |
W |
770 |
F |
0.30 |
| 123 |
631 |
K |
637 |
N |
0.30 |
| 124 |
643 |
R |
649 |
V |
0.29 |
| 125 |
846 |
G |
850 |
I |
0.29 |
| 126 |
647 |
I |
651 |
L |
0.29 |
| 127 |
649 |
V |
690 |
V |
0.29 |
| 128 |
670 |
W |
677 |
L |
0.29 |
| 129 |
852 |
N |
857 |
W |
0.29 |
| 130 |
861 |
W |
875 |
Y |
0.29 |
| 131 |
832 |
T |
836 |
V |
0.29 |
| 132 |
654 |
D |
658 |
T |
0.29 |
| 133 |
688 |
Y |
694 |
T |
0.28 |
| 134 |
626 |
S |
675 |
T |
0.28 |
| 135 |
723 |
S |
769 |
E |
0.28 |
| 136 |
796 |
T |
801 |
T |
0.28 |
| 137 |
838 |
L |
875 |
Y |
0.28 |
| 138 |
648 |
K |
693 |
L |
0.28 |
| 139 |
776 |
Y |
782 |
I |
0.28 |
| 140 |
770 |
F |
786 |
V |
0.28 |
| 141 |
746 |
V |
766 |
W |
0.28 |
| 142 |
816 |
V |
821 |
D |
0.28 |
| 143 |
730 |
W |
734 |
D |
0.28 |
| 144 |
686 |
V |
693 |
L |
0.28 |
| 145 |
690 |
V |
694 |
T |
0.28 |
| 146 |
726 |
G |
770 |
F |
0.27 |
| 147 |
645 |
T |
667 |
S |
0.27 |
| 148 |
721 |
T |
772 |
D |
0.27 |
| 149 |
685 |
Q |
694 |
T |
0.27 |
| 150 |
812 |
T |
818 |
K |
0.27 |
| 151 |
787 |
T |
798 |
I |
0.27 |
| 152 |
840 |
Q |
875 |
Y |
0.27 |
| 153 |
817 |
T |
821 |
D |
0.27 |
| 154 |
840 |
Q |
845 |
T |
0.27 |
| 155 |
629 |
V |
649 |
V |
0.27 |
| 156 |
633 |
W |
647 |
I |
0.27 |
| 157 |
861 |
W |
865 |
D |
0.27 |
| 158 |
818 |
K |
822 |
D |
0.27 |
| 159 |
851 |
L |
863 |
G |
0.27 |
| 160 |
851 |
L |
856 |
N |
0.27 |
| 161 |
633 |
W |
644 |
P |
0.27 |
| 162 |
773 |
L |
777 |
D |
0.27 |
| 163 |
629 |
V |
636 |
N |
0.27 |
| 164 |
651 |
L |
672 |
H |
0.26 |
| 165 |
757 |
T |
848 |
T |
0.26 |
| 166 |
760 |
V |
766 |
W |
0.26 |
| 167 |
761 |
T |
765 |
N |
0.26 |
| 168 |
836 |
V |
840 |
Q |
0.26 |
| 169 |
645 |
T |
649 |
V |
0.26 |
| 170 |
726 |
G |
802 |
T |
0.26 |
| 171 |
846 |
G |
853 |
E |
0.26 |
| 172 |
851 |
L |
857 |
W |
0.26 |
| 173 |
630 |
T |
635 |
D |
0.26 |
| 174 |
829 |
K |
834 |
I |
0.26 |
| 175 |
762 |
S |
853 |
E |
0.26 |
| 176 |
644 |
P |
649 |
V |
0.26 |
| 177 |
653 |
Q |
689 |
T |
0.26 |
| 178 |
838 |
L |
861 |
W |
0.26 |
| 179 |
846 |
G |
851 |
L |
0.25 |
| 180 |
820 |
W |
824 |
N |
0.25 |
| 181 |
842 |
G |
846 |
G |
0.25 |
| 182 |
860 |
T |
865 |
D |
0.25 |
| 183 |
708 |
M |
712 |
I |
0.25 |
| 184 |
701 |
T |
705 |
N |
0.25 |
| 185 |
834 |
I |
838 |
L |
0.25 |
| 186 |
849 |
A |
855 |
N |
0.25 |
| 187 |
829 |
K |
848 |
T |
0.25 |
| 188 |
845 |
T |
851 |
L |
0.25 |
| 189 |
853 |
E |
859 |
H |
0.25 |
| 190 |
631 |
K |
636 |
N |
0.25 |
| 191 |
649 |
V |
672 |
H |
0.25 |
| 192 |
724 |
I |
788 |
E |
0.25 |
| 193 |
830 |
R |
834 |
I |
0.25 |
| 194 |
841 |
D |
845 |
T |
0.25 |
| 195 |
730 |
W |
748 |
L |
0.25 |
| 196 |
721 |
T |
774 |
P |
0.25 |
| 197 |
647 |
I |
744 |
V |
0.25 |
| 198 |
685 |
Q |
695 |
K |
0.25 |
| 199 |
649 |
V |
653 |
Q |
0.25 |
| 200 |
774 |
P |
788 |
E |
0.25 |
| 201 |
628 |
T |
636 |
N |
0.25 |
| 202 |
856 |
N |
866 |
E |
0.25 |
| 203 |
644 |
P |
648 |
K |
0.25 |
| 204 |
623 |
G |
628 |
T |
0.24 |
| 205 |
661 |
T |
674 |
W |
0.24 |
| 206 |
629 |
V |
634 |
D |
0.24 |
| 207 |
848 |
T |
853 |
E |
0.24 |
| 208 |
845 |
T |
855 |
N |
0.24 |
| 209 |
843 |
K |
866 |
E |
0.24 |
| 210 |
665 |
N |
672 |
H |
0.24 |
| 211 |
859 |
H |
866 |
E |
0.24 |
| 212 |
652 |
Y |
658 |
T |
0.24 |
| 213 |
728 |
K |
803 |
I |
0.24 |
| 214 |
722 |
T |
776 |
Y |
0.24 |
| 215 |
653 |
Q |
678 |
D |
0.24 |
| 216 |
811 |
E |
818 |
K |
0.24 |
| 217 |
758 |
L |
766 |
W |
0.24 |
| 218 |
631 |
K |
635 |
D |
0.24 |
| 219 |
652 |
Y |
657 |
A |
0.24 |
| 220 |
816 |
V |
824 |
N |
0.24 |
| 221 |
838 |
L |
863 |
G |
0.24 |
| 222 |
843 |
K |
859 |
H |
0.24 |
| 223 |
649 |
V |
670 |
W |
0.24 |
| 224 |
829 |
K |
862 |
T |
0.24 |
| 225 |
851 |
L |
859 |
H |
0.24 |
| 226 |
833 |
E |
853 |
E |
0.24 |
| 227 |
651 |
L |
664 |
L |
0.24 |
| 228 |
859 |
H |
875 |
Y |
0.24 |
| 229 |
856 |
N |
861 |
W |
0.24 |
| 230 |
693 |
L |
698 |
G |
0.24 |
| 231 |
692 |
E |
696 |
V |
0.24 |
| 232 |
644 |
P |
696 |
V |
0.24 |
| 233 |
859 |
H |
865 |
D |
0.24 |
| 234 |
833 |
E |
850 |
I |
0.24 |
| 235 |
856 |
N |
863 |
G |
0.23 |
| 236 |
836 |
V |
866 |
E |
0.23 |
| 237 |
851 |
L |
861 |
W |
0.23 |
| 238 |
849 |
A |
853 |
E |
0.23 |
| 239 |
828 |
G |
832 |
T |
0.23 |
| 240 |
703 |
V |
707 |
D |
0.23 |
| 241 |
834 |
I |
839 |
Y |
0.23 |
| 242 |
724 |
I |
728 |
K |
0.23 |
| 243 |
866 |
E |
877 |
V |
0.23 |
| 244 |
858 |
T |
865 |
D |
0.23 |
| 245 |
792 |
K |
796 |
T |
0.23 |
| 246 |
636 |
N |
643 |
R |
0.23 |
| 247 |
629 |
V |
633 |
W |
0.23 |
| 248 |
837 |
E |
859 |
H |
0.23 |
Alignment robustness analysis
First most common residue correlation
Second most common residue correlation